Bestimmung der genomweiten exogenen Bindung von Pathogenen an das Wirtsgenom?
yahoo301503
Ich habe dieses Papier gelesen, in dem sie eine Region im Reisgenom spezifisch modifizieren, um die Bindungsstelle eines Pathogens, Xanthomonas oryzae , abzutragen und die Entführung eines Gennetzwerks im Reisgenom zum Vorteil des Pathogens zu unterbrechen.
Es ist ein interessantes Konzept, aber ich habe mich gefragt, wie man die Bindung dieser Pathogen-Effektoren an den Wirt de novo bestimmen würde, wenn man sie nicht kennt. Was wären die Schritte, um zu bestimmen, wo die TFs des Pathogens an das Wirtsgenom binden?
Bearbeiten: Es besteht die Möglichkeit, nicht zu wissen, welche Proteine an das Wirtsgenom binden. Wie würden also in diesem Fall die Antikörper entwickelt?
Bezug:
Antworten (2)
Konrad Rudolf
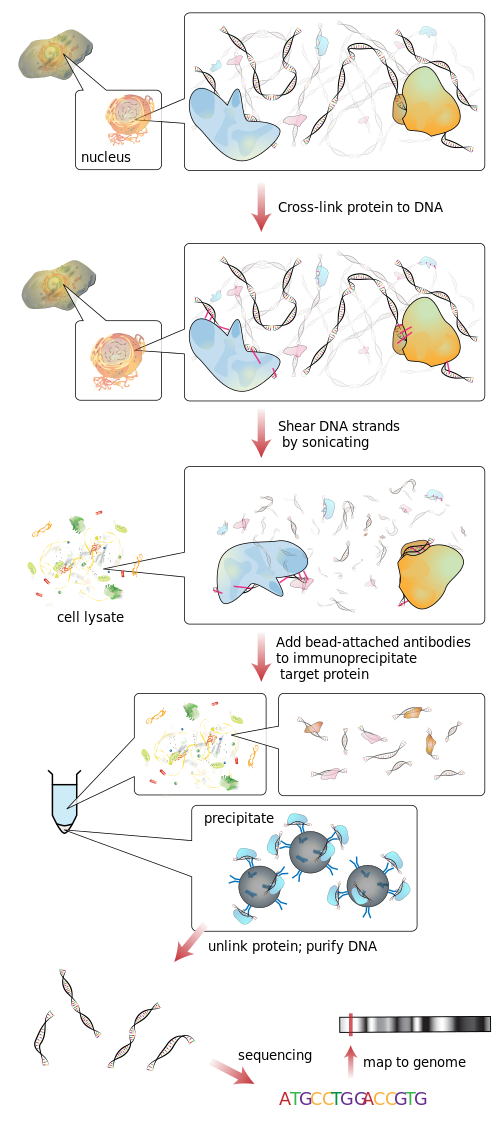
Die dafür verwendeten Techniken sind ChIP-seq und ChIP-chip.
Im Grunde Sie
- Lassen Sie den Erreger an die (hochreplizierte) DNA binden
- Zerschneiden Sie die DNA in kleine zufällige Stücke durch Beschallung
- Anreicherung ("pull down") der erregergebundenen DNA-Fragmente durch Verwendung eines bekannten Antikörpers, der an den Erreger bindet
- Sequenzieren Sie die so angereicherte DNA
- Kartieren Sie die sequenzierten Fragmente zurück zu ihrem ursprünglichen Ort im Genom.
Wikipedia hat ein schönes Bild, das diesen Vorgang etwas detaillierter erklärt:
Daniel Steh
Ein Kollege von mir entdeckte die Chiffre, die die TAL-Effektor-DNA-Spezifitäten bestimmt, die in diesem kurzen Artikel beschrieben wird . Diese Spezifitäten wurden bestimmt, indem an DNA gebundene TAL-Effektoren beobachtet und aufgezeichnet wurden, wie oft ein bestimmter Repeat-Variable-Direst (RVD) einem bestimmten Nukleotid entsprechen würde (unter Verwendung einer Gewichtsmatrix).
Nachdem nun die Spezifitäten bestimmt wurden, besteht die Identifizierung der TAL-Effektor-Zielstellen einfach darin, die Wahrscheinlichkeit zu messen, dass sich die RVD-Sequenz dieses TAL-Effektors an einer neuen Sequenz ausrichtet. Einige Anwendungen werden hier und hier beschrieben .
Der TALENT 2.0-Server (eingeladenes Papier wird gerade begutachtet) bietet Tools zum Entwerfen benutzerdefinierter TAL-Effektoren, die auf spezifische DNA-Sequenzen abzielen, oder zum Identifizieren potenzieller Off-Target-Bindungsstellen in einer bestimmten Sequenz oder einem bestimmten Genom.
Verweise:
Wie können Promotorbindungsstellen bestimmt werden?
Wie stark beeinflusst der Abstand zwischen einer Transkriptionsfaktor-Bindungsstelle und einem Promotor die Transkription?
Identifizierung, welche SNPs in TFBS (Hefe) sitzen
Was macht DNA-Sequenzen aus biologischer Sicht am unterschiedlichsten/erkennbarsten? [Duplikat]
Regulatorische Elemente finden: Wie haben die Leute das gemacht?
Lokalisierung von TFBS im Genom
Anzahl der Transkriptionsfaktor-Gene im menschlichen Genom
An welche anderen Stellen außer allosterischen Stellen binden nicht-kompetitive Inhibitoren?
Alle Körperzellen enthalten das gleiche Genom, woher wissen sie dann, dass sie sich zu einem bestimmten Organ entwickeln sollen?
Gen-Silencing: relative oder absolute Anzahl methylierter CpG-Stellen?